오늘의 논문

이번 논문은 DeepSEED : 3D Squeeze-and-Excitation Encoder-Decoder Convolutional Neural Networks for Pulmonary Nodule Detection 입니다.
https://ieeexplore.ieee.org/stamp/stamp.jsp?tp=&arnumber=9098317
DeepSEED: 3D Squeeze-and-Excitation Encoder-Decoder Convolutional Neural Networks for Pulmonary Nodule Detection
Pulmonary nodule detection plays an important role in lung cancer screening with low-dose computed tomography (CT) scans. It remains challenging to build nodule detection deep learning models with good generalization performance due to unbalanced positive
ieeexplore.ieee.org
여기서 논문을 바로 보실 수 있습니다.
논문 목차
1. Introduction
2. Methods
2.1 Network Structure
2.2 Classification and Regression Loss Functions
3. Experimental Results
3.1 Datasets
3.2 Implementation Details
3.3 Performance of Different Methods under Comparison
3.4 Ablation studies
4. Conclusions
1. Introduction
DeepSEED에서는 1-stage object detection 방식을 사용하였고 기존 3D RPN에 squeeze-and-excitation structure, positive와 negative sample들 간의 imbalance 문제를 해결하기 위해서 Focal Loss를 도입했습니다. SOTA와 성능 비교를 하기 위해서 LUNA16과 LUNA16보다 slice thickness가 큰 LIDC/IDRI에서 실험결과를 측정했습니다.
2. Methods
2.1 Network Structure

● 3D RPN backbone
● backbone은 ResNet-18 structure를 encoder에 뒀음
● Decoder는 3개의 layer로 구성되어있고 residual block과 skip connection이 있습니다.
● 각각의 residual block은 Squeeze-and-Excitation을 포함하고 있습니다.
RPN의 output은 각각의 anchor의 classification probability score p와 nodule의 spatial information인 x, y, z, r입니다.
Squeeze-and-Excitation (SE)

Squeeze-and-Excitation (SE) block은 Global Average Pooling을 통해서 channel 수의 길이를 가진 1차원 텐서로 만들고 Fully connected Layer - RELU - Fully connected Layer - Sigmoid를 거치는 연산입니다. 이 과정의 의미는 각 채널별로 가중치를 부여하는 건데 더 중요한 정보를 가지고 있는 채널의 가중치가 높아지게 됩니다. Classification Task에서 SE block을 추가하게 된다면 계산복잡도를 거의 증가시키지 않으면서 성능 향상을 줄 수 있다고 합니다.
자세한 설명은 아래 링크를 참조해주세요.
https://jayhey.github.io/deep%20learning/2018/07/18/SENet/
SENet(Squeeze and excitation networks)
2017 이미지넷 챌린지(ILSVRC 2017)에서 우승한 SENet입니다. 피쳐맵의 압축(squeeze)과 재조정(recalibration)을 통한 스케일이 핵심인 방법론입니다.
jayhey.github.io
2.2 Classification and Regression Loss Fuctions
RPN에서는 positive, negative nodule을 구별하기 위해서 Classification Loss를 사용하고, BoundingBOX를 예측하기 위해서 Regression Loss를 사용합니다. positive, negative sample의 imbalance를 해결하기 위해서 Focal Loss를 도입하였습니다.
$$ L_{cls} = -\alpha(1 - p_t)^{\gamma} log(p_t) $$
본 논문에서는 $\alpha = 0.5, \gamma = 2$가 사용되었습니다.
$$ L_{reg} = \sum_{k} S(G_k, P_k) $$
S()는 smoothed L1 function이고
$ G_k = \left( \frac{x_g-x_a}{r_a}, \frac{y_g-y_a}{r_a}, \frac{z_g-z_a}{r_a}, log \left( \frac{r_g}{r_a} \right) \right) $
$ P_k = \left( \frac{x-x_a}{r_a}, \frac{y-y_a}{r_a}, \frac{z-z_a}{r_a}, log \left( \frac{r}{r_a} \right) \right) $
*$ x_a, y_a, z_a, r_a $는 anchor의 location과 radius
Total Loss function : $ L = L_{cls} + p^{*} L_{reg} $
* positive sample일 때 $p^{*}=1$이고, negative sample일 때 $p^{*}=0$ 가 됩니다.
3. Experimental Results
3.1 Datasets
● LIDC/IDRI와 LUNA16을 평가하기 위한 데이터셋으로 사용했습니다.
● 두 데이터셋 모두 3mm보다 큰 nodule이 label되어 있습니다.
● LUNA16 데이터셋은 LIDC/IDRI에서 slice thickness가 2.5mm보다 큰 것을 제외하였고, 888 CT scans와 1186개의 labeled nodule이 있습니다.
● LUNA16에서 10-fold cross-validation을 평가하였고, LIDC/IDRI에서 LUNA16을 제외한 122 subject를 테스트셋으로 평가하였습니다.
| LUNA16 | LIDC/IDRI | |
| mean | 8.3mm | 4.8mm |
| variance | 12.8mm | 10.6mm |
3.2 Implementation Details
● 폐를 분할하기 위해 binary segmentation으로 전처리 [-1200, 600]으로 각 pixel 값을 clip
● [-1200, 600]으로 각 pixel 값을 clip 후 [0, 1]로 normalize
● 128×128×128 크기로 랜덤하게 crop하여 input
● data augmentation으로 random flipping, 0.75~1.25 scaling 사용
● IoU > 0.5 : positive sample, IoU < 0.02 : negative sample
● anchor size : [5, 10, 20]
● 테스트 과정에서는 image patch를 208×208×208로 해서 각 patch마다 32 pixel만큼 겹치게 함
평가지표로 FROC를 사용하였습니다.
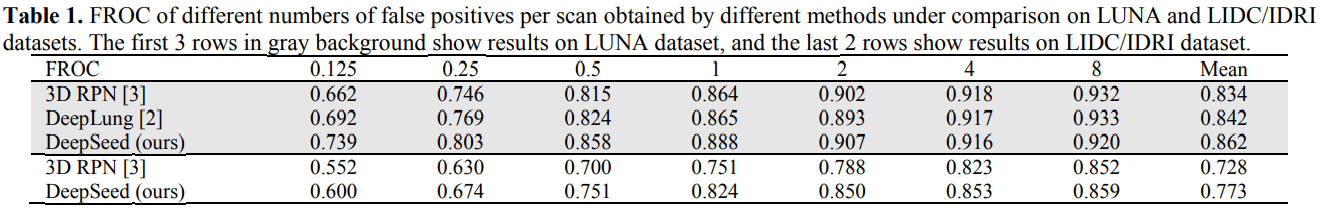
→ FROC는 LUNA16에서 제공한 평가지표인데 average number of false positives per scan별로 sensitivity를 측정하게 됩니다. 전체 점수는 0.125, 0.25, 0.5, 1, 2, 4, 8 각각의 false positives per scan별로 sensitivity를 측정해서 평균을 냅니다.
3.3 Performance of Different Methods under Comparison

DeepSEED는 LUNA16에서 FROC score 86.2%, LIDC/IDRI에서 77.3%를 달성하였습니다.
특히 DeepSEED는 SOTA nodule detection 방법보다 false positives per scan 0.125~2에서 각각 4.1%, 3.0% 능가했습니다.
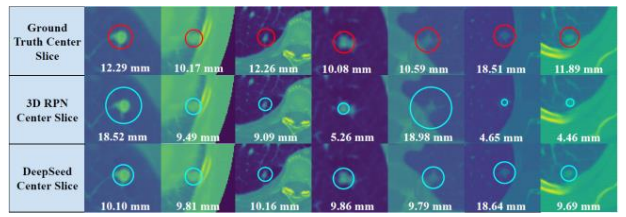
위의 Figure는 BoundingBOX 크기를 예측하는 측면에서 DeepSEED와 3D RPN을 비교한 것입니다.
DeepSEED는 기존의 3D RPN보다 정확한 BoundingBOX 크기를 예측했습니다.
3.4 Ablation studies

method의 어떤 component가 성능 향상에 기여하는지 확인하기 위해서 3D RPN, DeepSEED, DeepSEED without FocalLoss, DeepSEED without SE blocks을 비교했습니다.
→ 위 실험 결과로 SE sturcture와 focal loss는 3D RPN의 성능 향상에 기여한 것을 볼 수 있습니다.
4. Conclusions
본 논문에서 저자들은 새로운 End-to-End 3D deep learning network를 제안하였습니다. sample imbalance와 false positive rate 문제를 해결하기 위해서 Focal classification Loss를 사용하였습니다. LUNA16과 LIDC/IDRI에서 SOTA인 네트워크보다 성능이 향상되었고, ablation study를 통해 Squeeze-and-Excitation structure와 focal classification loss가 nodule detection 성능 향상에 중요하다는 것을 보여주었습니다.
'논문 리뷰' 카테고리의 다른 글
| [논문 리뷰] End-to-end Lung Nodule Detection in Computed Tomography (0) | 2022.08.11 |
|---|---|
| [논문 리뷰]OpenPose: Realtime Multi-Person 2D PoseEstimation using Part Affinity Fields (0) | 2021.07.04 |


댓글